第一种;直接使用提供的BSCMatrixx进行运行;
第二种;如果习惯使用cellranger,可以使用提供的FQ_BMKMANU_to_10X 模块将BMKMANU DG1000的数据转换为10X格式直接使用cellranger分析
策略一
直接使用BSCMatrix进行分析
1、软件依赖与安装
1).Python: 3.8及以上,需要安装模块: plotly lz4 numpy Cython h5py scipy pandas pytables sklearn
2).R: 4.0及以上,需要安装包: Seurat dplyr tibble ggplot2 plotly htmlwidgets kableExtra htmltools shiny knitr rmarkdown optparse
3).Cell Ranger: 7.0及以上
4).Seqkit (https://bioinf.shenwei.me/seqkit/download/)
依赖的软件需要使用export添加到环境变量中,以实现流程的调用。
2、数据准备
1).测序数据,双端FASTQ数据
2).参考基因组数据,基因组序列文件和gtf文件(配置文件使用的注释文件需要包含gene和exon)
3、配置文件填写
### Globle parameters
## data 测序数据
FQ1 /path/to/read_1.fastq
FQ2 /path/to/read_2.fastq
## Ref genome 参考基因组和gtf文件
FA /path/to/ref/file
GFF /path/to/gtf/file
## out put 输出路径以及输出结果前缀
OUTDIR /path/to/result/dir/
PREFIX outfile-prefix
##other parameters 其它配置参数(期望细胞数、线程数目、内存上限100Gb,Nobam?设置为0代表不输出bam,设置其它字符代表输出)
EC 3000
Threads 8
RAM 100
Nobam? 1
ENV ###R和python解释器路径(可选,如果不提供,则放到环境变量中)
Rscript /path/to/R/bin/
PYTHON /path/to/python/bin/
4、流程运行
1).流程说明
流程分为4个步骤,功能如下:
1.1步骤1,运行fastq2BcUmiSC_v1.1,识别fastq数据中的barcode、umi
1.2步骤2,调用Cell Ranger程序,获取基因表达矩阵
1.3步骤3,运行QC,进行umap和tsne分析
1.4步骤4,运行WebReport,得到网页版报告
2).流程参数
-c config.txt 配置文件
-s 步骤选择,0为运行1-4所有步骤,也可选择个别步骤单独运行,多个步骤中间使用”,”分割。
3).参考命令
./BSCMatrix -c config.txt -s 0
./BSCMatrix -c config.txt -s 1,2,3,4
./BSCMatrix -c config.txt -s 1,2
5、结果说明
01.fastq2BcUmiSC ##步骤1运行结果目录
├── prefix.bc_stat ##不同barcode统计
├── prefix.filter ##barcode 过滤信息
├── prefix.qual.stat ##Reads 统计
├── prefix.stat ##barcode检测统计
└── prefix.umi ##Reads barcode umi信息
02.cellranger/ ##步骤2 运行结果目录
├── bmk_10x_barcode.xls ## barcode对照表
├── data/ ##数据存放目录
├── INDEX/ ##参考基因组索引文件夹
├── Log.out ##索引构建日志文件
├── R1.fq.gz ## R1端数据
├── R2.fq.gz ## R2 端数据
├── ReadID.xls ##ReadID 信息
└── prefix/ ##调用Cell Ranger 软件分析结果目录
03.cluster/ ## 步骤3 运行结果目录
├── cluster.csv ##细胞聚类结果
├── marker_gene.csv ##marker gene
├── tsne_df.xls ##tsne聚类结果
├── tsne_files/ ##tsne html附录文件
├── tsne.html ##tsne html 格式图片
├── tsne.pdf ##tsne pdf格式图片
├── umap_df.xls ##umap 聚类结果
├── umap_files/ ##umap html附录文件
├── umap.html ##umap html格式图片
└── umap.pdf ##umap pdf格式图片
04.WebReport/ ##步骤4 运行结果目录
├── 10x ##调用Cell Ranger程序执行的结果
└── bmk ##网页报告
##############BSCMatrix 环境依赖######################################
1、conda 安装
#下载
wget https://repo.anaconda.com/miniconda/Miniconda3-py39_4.12.0-Linux-x86_64.sh
#下载完成之后运行(按提示安装)
sh Miniconda3-py39_4.12.0-Linux-x86_64.sh
#安装完成后执行以下命令
source ~/.bashrc
2、conda 环境配置
conda create -n (环境名) python=3.9
#激活创建的环境
conda activate (环境名)
#添加镜像源
conda config –add channels https://mirrors.bfsu.edu.cn/anaconda/cloud/bioconda/
conda config –add channels https://mirrors.bfsu.edu.cn/anaconda/cloud/conda-forge/
conda config –add channels https://mirrors.bfsu.edu.cn/anaconda/pkgs/free/
conda config –add channels https://mirrors.bfsu.edu.cn/anaconda/pkgs/main/
#查看镜像源
conda config –show-sources
3、安装python模块
pip3 install -i https://pypi.tuna.tsinghua.edu.cn/simple plotly
pip3 install -i https://pypi.tuna.tsinghua.edu.cn/simple lz4
pip3 install -i https://pypi.tuna.tsinghua.edu.cn/simple Cython
pip3 install -i https://pypi.tuna.tsinghua.edu.cn/simple h5py
pip3 install -i https://pypi.tuna.tsinghua.edu.cn/simple scipy
pip3 install -i https://pypi.tuna.tsinghua.edu.cn/simple tables
pip3 install -i https://pypi.tuna.tsinghua.edu.cn/simple sklearn
pip3 install -i https://pypi.tuna.tsinghua.edu.cn/simple pandas
4、安装指定R版本(v4.2)
conda install -c conda-forge r-base=4.2
5、安装R包
conda install -c conda-forge r-seurat=4.3.0
conda install -c conda-forge r-dplyr=1.1.0
conda install -c conda-forge r-tibble=3.1.8
conda install -c conda-forge r-plotly=4.10.1
conda install -c conda-forge r-htmlwidgets=1.6.1
conda install -c conda-forge r-kableextra=1.3.4
conda install -c conda-forge r-htmltools=0.5.4
conda install -c conda-forge r-shiny=1.7.4
conda install -c conda-forge r-knitr=1.42
conda install -c conda-forge r-rmarkdown=2.20
conda install -c conda-forge r-optparse=1.7.3
6、Cell Ranger安装
#下载
wget -O cellranger-7.1.0.tar.gz “https://cf.10xgenomics.com/releases/cell-exp/cellranger-7.1.0.tar.gz?Expires=1675964753&Policy=eyJTdGF0ZW1lbnQiOlt7IlJlc291cmNlIjoiaHR0cHM6Ly9jZi4xMHhnZW5vbWljcy5jb20vcmVsZWFzZXMvY2VsbC1leHAvY2VsbHJhbmdlci03LjEuMC50YXIuZ3oiLCJDb25kaXRpb24iOnsiRGF0ZUxlc3NUaGFuIjp7IkFXUzpFcG9jaFRpbWUiOjE2NzU5NjQ3NTN9fX1dfQ__&Signature=IFr7ONDqEkZRR6QpU~A6719a9Mc2SD2tI1z6RrGldFFTCiY6Z7VR0x0Gr90jtvTUmYTJ2S0NyuK6SVmdeIZUCcbjz9elG1ImGx7AprTCRD3m~0se-xha2lFr87bEsbAa-7uoyW14wXRlj17b0oG9WomNvVSNNKJSzSSfCkqX3Ev9B82b~DMD-7-Hlb8lAsorv18R8y41T4UihIRdY-LE-I5Gk3fTodmBUjvSEuI3VEalsrVsrN5AdBDpwiCPqSiExODVM0RIsUDV158ceAYFiu5Y9wgbwQVOFMZGYI0d-6tO1VPo4RwwWl0X7c2q21im6BNSQrhQzoDv5cj5COesmQ__&Key-Pair-Id=APKAI7S6A5RYOXBWRPDA”
##下载完成之后解压
tar -zxvf cellranger-7.1.0.tar.gz
##配置环境变量
echo “PATH=安装路径/cellranger/7.1.0/bin:$PATH” >~/.bashrc
7、安装seqkit
conda install -c bioconda seqkit
策略二
使用FQ_BMKMANU_to_10X 对原始数据进行转换,然后使用cellranger进行分析
#########################软件说明#########################################
./dealFQ
Description:
Version:v1.0
Usage:
-r1 Read1 Fastq must be given #########R1端数据
-r2 Read2 Fastq must be given #########R2端数据
-o outdir optional [./] ########输出路径
-k keyword optional [bmk] #########关键字
-t threads optional [4] ##########线程数目
-h help document
Example:
dealFQ -r1 R1.fq.gz -r2 R2.fq.gz -o analysis -k bmk -t 8
#########################结果说明##########################################
├── 01.fastq2BcUmiSC
│?? ├── *.bc_stat ##不同barcode统计
│?? ├── *.filter ##barcode 过滤信息
│?? ├── *.qual.stat ##Reads 统计
│?? ├── *.stat ##barcode检测统计
│?? └── *.umi ##Reads barcode umi信息
└── 02.data
├── bmk_10x_barcode.xls ## barcode对照表
├── data
│?? ├── *_S1_L001_R1_001.fastq.gz ##cellranger分析所需数据 (R1.fq.gz的硬链接)
│?? └── *_S1_L001_R2_001.fastq.gz ##cellranger分析所需数据(R2.fq.gz的硬链接)
├── R1.fq.gz
├── R2.fq.gz
└── ReadID.xls ##ReadID 信息
######################################
可以使用软件dealFQ 将DG1000数据转换成10x cellranger兼容数据,然后用cellranger进行下游分析,文件夹内02.data/data数据即是cellranger分析所需数据。
目前常见的捕获单细胞技术方法有:微流控和微孔板法。本文通过基于微流控法的平台-百创DG1000来精准捕获细胞,并进行后续的逆转录、cDNA合成与扩增、文库构建等实验,从而获得每个细胞的转录组信息。
1、芯片加样
处理好的细胞悬液尽量在30min以内上机实验,长时间放置,会导致细胞活性降低。按照一定的加样顺序,分别将细胞相、胶珠相(DG1000-3′ gel beads)、油相加入到芯片(图1:左图)对应孔中,然后将芯片放入到百创DG1000微液滴生成仪中(图1:右图),启动开关。

图1:百创C1000芯片结构(左图);百创DG1000微液滴生成仪(右图)
2、形成GEMs
仪器运行后,通过控制流速的方法,使得在“Y型”接口处,一个胶珠会吸附单个细胞及逆转录预混液一起向右流动。到了“十字”接口处,它们与油相接触后,每一个油滴中会包裹着一个胶珠与一个细胞,也就是“油包水”(GEMs)结构。

图2:形成“油包水”的原理(左);“油包水”(GEMs)结构示意图(右)。
引物结构
- 百创DG1000-3′ gel beads(胶珠)上固定的引物由4部分组成 :Read1、Cell Barcode、UMI、Poly(dT)VN。
- Read 1: 是一段已知的短核昔酸序列,主要用于后续的上机测序
- Cell Barcode:片段长度54bp,种类约为88万种,一个胶珠对应于一个特异Cell Barcode序列。
- UMI:在 mRNA反转录后,每一个mRNA随机连上一个UMI,可以通过计数不同的UMI,最终确定mRNA的数量。
- Polv(dT)VN:与mRNA3’端的poly(A)结合,进行后续的逆转录反应
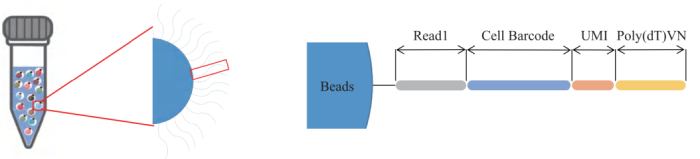
图3:引物结构示意图。
3、cDNA生成与扩增
GEMs形成后,细胞裂解释放出mRNA;同时胶珠也会自动溶解,释放出大量含有Barcode序列的引物,并利用引物末端的Ploy(dT)VN序列捕获液滴中的mRNA,并在一个个GEM中独立完成mRNA逆转录,产生带有Barcode和UMI信息的cDNA。随后“油包水”结构裂开,进行cDNA扩增。
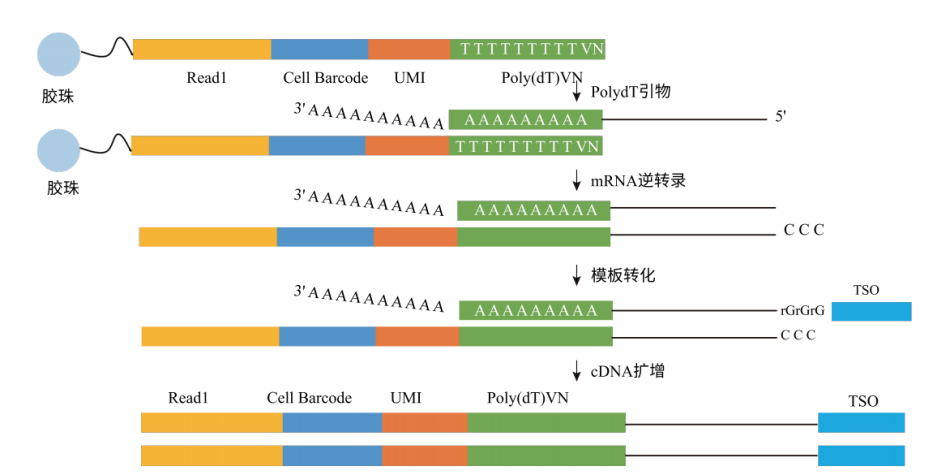
将扩增得到的cDNA纯化后,可用于后续构建测序文库。由于每一个单细胞的cDNA文库标记上了细胞条形码(Barcode)。测序时,就把携带相同Barcode的序列视为来自同一个细胞,达到区分细胞的目的。
以上就是生成GEMs、mRNA逆转录、以及cDNA合成与扩增的全部内容。下期会具体介绍数据分析指标的意义。
单细胞平台-百创DG1000
百创DG1000是基于微流控、油滴包裹和Barcode标记等技术来实现高通量的单细胞精准捕获。除了仪器以外,百创DG1000还有配套的2×4的独立芯片及配套试剂盒。
百创DG1000部分数据展示
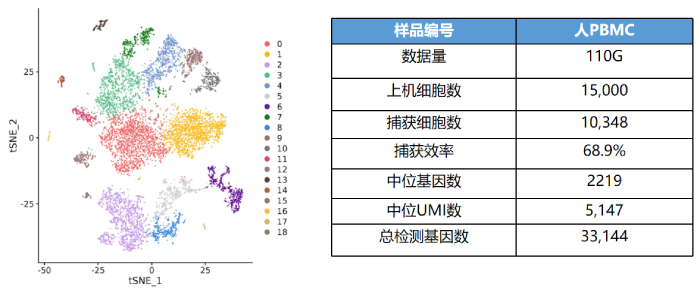
人PBMC
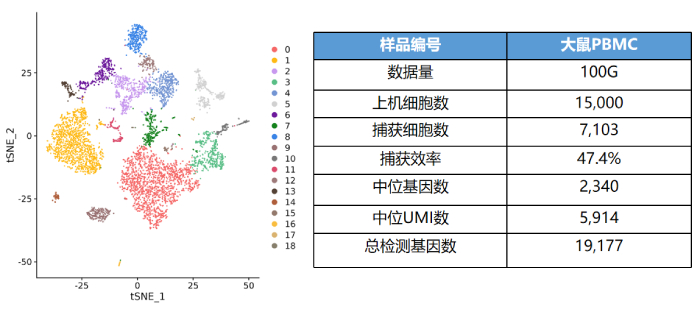
大鼠PBMC

鸡PBMC
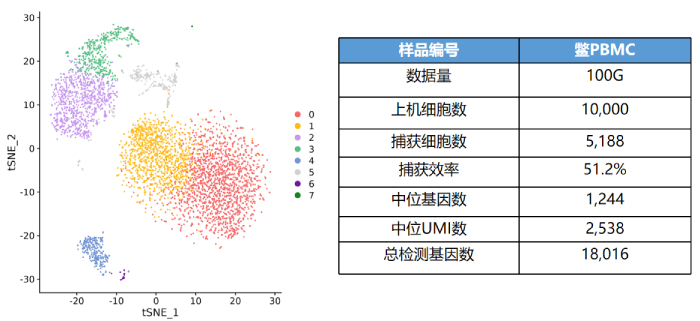
鳖PBMC

黄颡鱼PBMC

牡蛎PBMC

小鼠胚胎
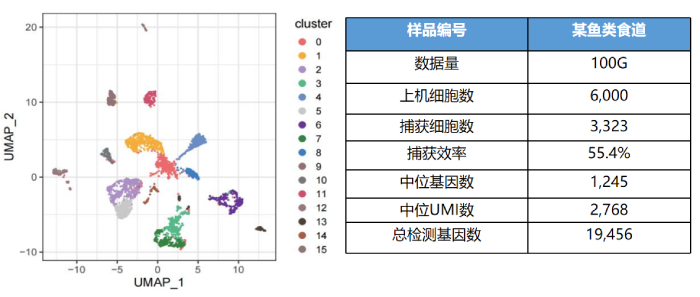
某鱼类食道
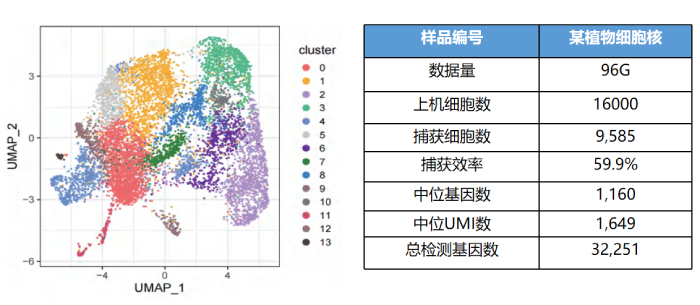
某植物细胞核
如果您对百创DG1000单细胞平台感兴趣,欢迎点击下方按钮联系我们,我们将免费为您设计文章思路方案。
]]>
10月27日,昆明动物研究所基因组研究中心和北京百迈客生物科技有限公司(以下简称“百迈客”)宣布达成战略合作,在昆明动物所举办了合作签约仪式。由青岛百创智能制造技术有限公司(以下简称“百创智造”)自主研发的单细胞实验平台百创DG1000正式投放至昆明动物所基因组研究中心,双方本着平台共享、经验共享的原则达成后续深入合作。基因组研究中心主任王国栋研究员与百创智造副总裁刘敏及百迈客农学事业部总经理王淑英就单细胞未来发展方向及应用做了深入交流,并达成了重要战略合作。此次合作将推动单细胞研究在动物领域的发展,让单细胞技术能够惠及更多的科研群体,同时也推动百迈客科研进步、技术革新。

昆明动物所生物多样性基因组中心主任王国栋(右三),百创智造副总裁刘敏(正中),百迈客农学事业部总经理王淑英(左三)

百创智造副总裁刘敏对本项目进行介绍
百创智造副总裁刘敏表示:百创智造秉承“为生命科技研究提供更多可能”的理念,旨在创新开发仪器,芯片和试剂的同时,为实现生命科技核心仪器及配套试剂耗材的国产自主化贡献力量。历时3年潜心研发,于今年推出了单细胞实验平台百创DG1000和空间转录组芯片百创S1000,本次很荣幸能在昆明动物研究所投放DG1000仪器,希望在我们双方共同合作下,助力科研工作者开启时空转录组研究时代的同时,逐渐提升国产平台在生物技术研究领域的核心价值。
百迈客农学事业部总经理王淑英表示:目前我们百创DG1000单细胞实验平台现已完成人、多种动植物和微生物等样本的科研合作项目百余个,积累了丰富的项目经验;本项目旨在将百迈客提供自主研发单细胞实验仪器DG1000落户昆明动物所基因组实验中心,借助贵单位的实验平台共建单细胞实验技术联合实验室,进一步提升百迈客自主研发平台在时空组学研究领域的认可度,同时也希望能给贵单位的科研工作者提供更便捷高效的服务,最终实现互惠互利。

实验人员安装及开始测试百创DG1000仪器
百创DG1000仪器介绍
单细胞极速微液滴系统百创DG1000,利用微流控、油滴包裹和Barcode标记等技术来实现高通量的细胞捕获,包含微液滴生成平台,成套的试剂芯片耗材,提供从原始下机数据到基因表达量矩阵构建的开源软件。

图4 百创DG1000产品组成
百创DG1000技术流程
百创DG1000利用Droplet技术来实现高通量的细胞捕获。能够一次性分离、并标记500–10000个单细胞,从而获得每个细胞的转录组信息。
细胞与胶珠分别从Y型流道出来后经过油相生成GEMs,仅在2 min内可生成15-20W皮升级微液滴。
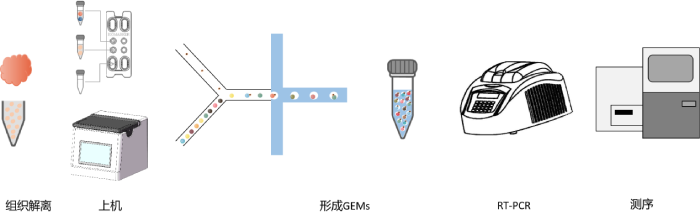
图5 百创DG1000技术流程图
百创DG1000产品优势

百迈客单细胞测序技术服务介绍
百迈客作为为数不多的同时拥有单细胞测序国内外双平台的公司,拥有丰富的单细胞空间多组学项目经验。现已累计100+物种、制备200+种组织类型单细胞样本。百迈客基于大量的动植物单细胞检测技术经验,已经建立成熟的单细胞悬液和植物单细胞核悬液制备体系,搭建了完善的分析流程。
百迈客开展的单细胞测序项目,不仅在医学领域蓬勃发展,农学领域也迎头赶上。项目经验覆盖人、鼠、畜牧类动物、水生动物、植物等,组织类型有分选的PBMC,肝、肺、胚胎、肠、耳蜗、皮肤、胃,植物的叶片、花、芽、根尖、幼果、幼茎等。无论简单还是复杂的组织类型,百迈客都坚持创新、勇于挑战、迎难而上!
百迈客单细胞部分项目案例

(仅展示近期项目,更多详情请点击下方按钮联系我们获取)
2022 CBIC第四届细胞生物产业大会、第二届中国生物医药创新合作大会于2022年7月28-29日在北京隆重举行。青岛百创智能制造技术有限公司(百创智造)受邀参会。百创智造刘敏副总裁以“把薪助火 || 百万级单细胞、亚细胞级分辨率空间转录组”进行了主题演讲,分别以单细胞测序助力靶药研究、单细胞eQTL助力疾病医疗、时空组学指导临床用药等视角与众多参会代表分享、探讨了时空组学在精准医疗领域的研究进展与未来展望。

图1 刘敏副总裁发表主题演讲
图2 刘敏副总裁和参会代表分享单细胞在医疗领域的研究进展
同时,刘敏副总裁也分享了百创智造助力时空组学向临床医疗迈进的亚细胞级空间转录组芯片-百创S1000和单细胞微液滴平台-百创DG1000。通过与来自全国各地参会代表的沟通和交流,参会代表对我们百创S1000可调节的亚细胞分辨率芯片和百创DG1000极快的单细胞微液滴系统产生了浓厚的兴趣,并希望能够进一步探讨和合作。
会议期间,到访我们展台的代表络绎不绝。通过本次会议,我们也对国内细胞生物研究在精准医学的发展有了新的理解,对单细胞多组学在临床应用有了更加明确的方向。这些也为我们日后的相关工作提出了指导意见。

图3 参会代表了解百创S1000和百创DG1000
百创智造单细胞平台及配套试剂
为了突破单细胞试剂耗材及仪器长期以来的国外技术垄断,降低单细胞应用的成本,让单细胞技术能够惠及更多的科研群体,经过多年潜心研发,百创智造发布自主研发的极速单细胞微液滴系统-百创DG1000。百创DG1000是基于微流控、油滴包裹和Barcode标记等技术来实现高通量的细胞捕获。除了仪器以外,百创DG1000还有配套的2×4的独立芯片及配套试剂盒(图4)。

图4百创DG1000
百创DG1000技术特点:
- 轻巧便携:背包即走,携带方便,便于实地取样,应用场景更广;
- 极速:110S即可完成整个反应,减少环境对细胞状态的应激影响;
- 芯片使用灵活:芯片2*4独立设计,1~8样本灵活上机;
- 兼容更大的细胞直径:已成功支持到细胞直径60μm。
PBMC重复性检测:将相同的PBMC细胞悬液分3份,通过3个不同实验人员,用3台百创DG1000仪器,独立进行单细胞测序,得到了几乎一致的细胞聚类分群结果,证明百创DG1000具有很好的可重复性及稳定性(图5)。
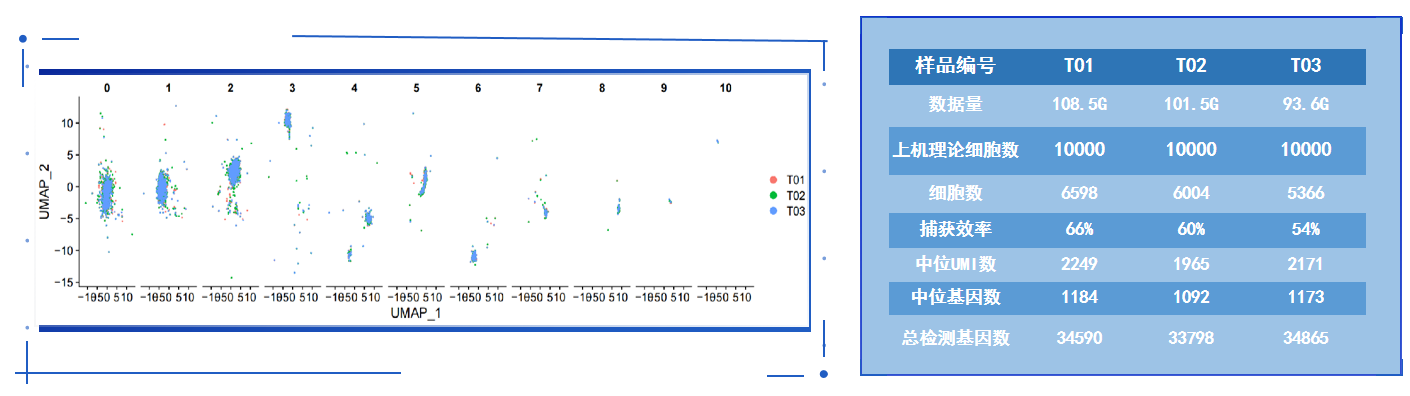
图5 PBMC重复性测试数据
百创智造空间转录组芯片与配套试剂
为了打破空间测序市场被国外公司长期占据,以及改变主流商业化平台的分辨率较低的的局面,经过多年的潜心研发,百创智造发布了基于微孔微珠的亚细胞级分辨率的空间转录芯片及配套试剂-百创S1000。该技术直接将空间组学分辨率从100μm(主流技术平台)提高到~5μm的亚细胞水平,标志着全面迈入国产化的空间转录组亚细胞技术时代的到来(图6)。

图6 百创S1000
百创S1000技术特点:
- 亚细胞级分辨率:分辨率可达到5μm;
- 1~8捕获区域:可灵活定制芯片、节省时间和实验成本;
- 支持HE染色:更大程度上还原组织病例形态;
- 支持多级分辨率分析:可100μm?~5μm灵活调整。
大鼠胚胎亚细胞分辨率实测数据展示:与100μm分辨率(主流技术平台)对比,高分辨率(~50μm)下HE染色组织形态学与spots聚类空间分布图重合度更高,精准识别空间基因表达的异质性和组织区域的特异性(图7)。
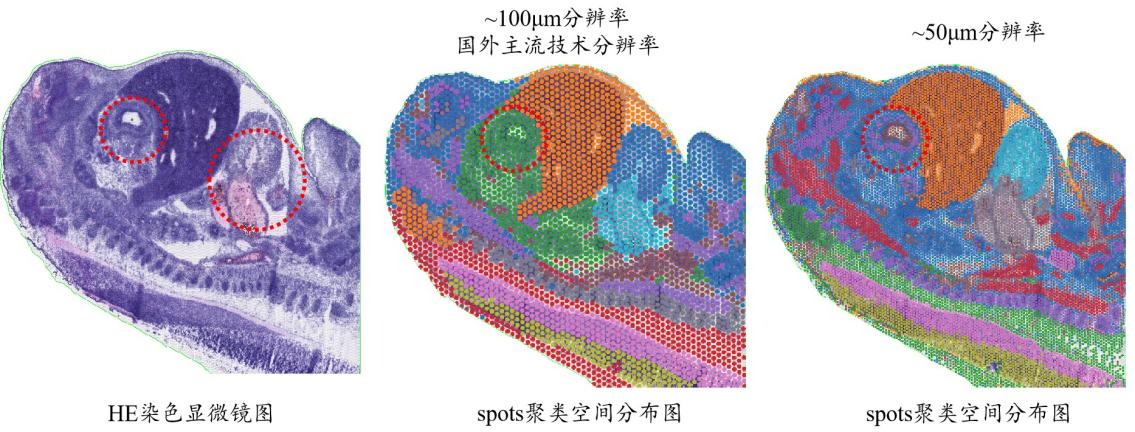
图7:百创S1000胚胎亚细胞分辨率空间转录组检测结果

扫码观看CBIC北京细胞生物医药产业大会报告视频回放
如果您对我们的平台感兴趣,欢迎点击下方联系我们。
]]>


